OpenMed
综合介绍
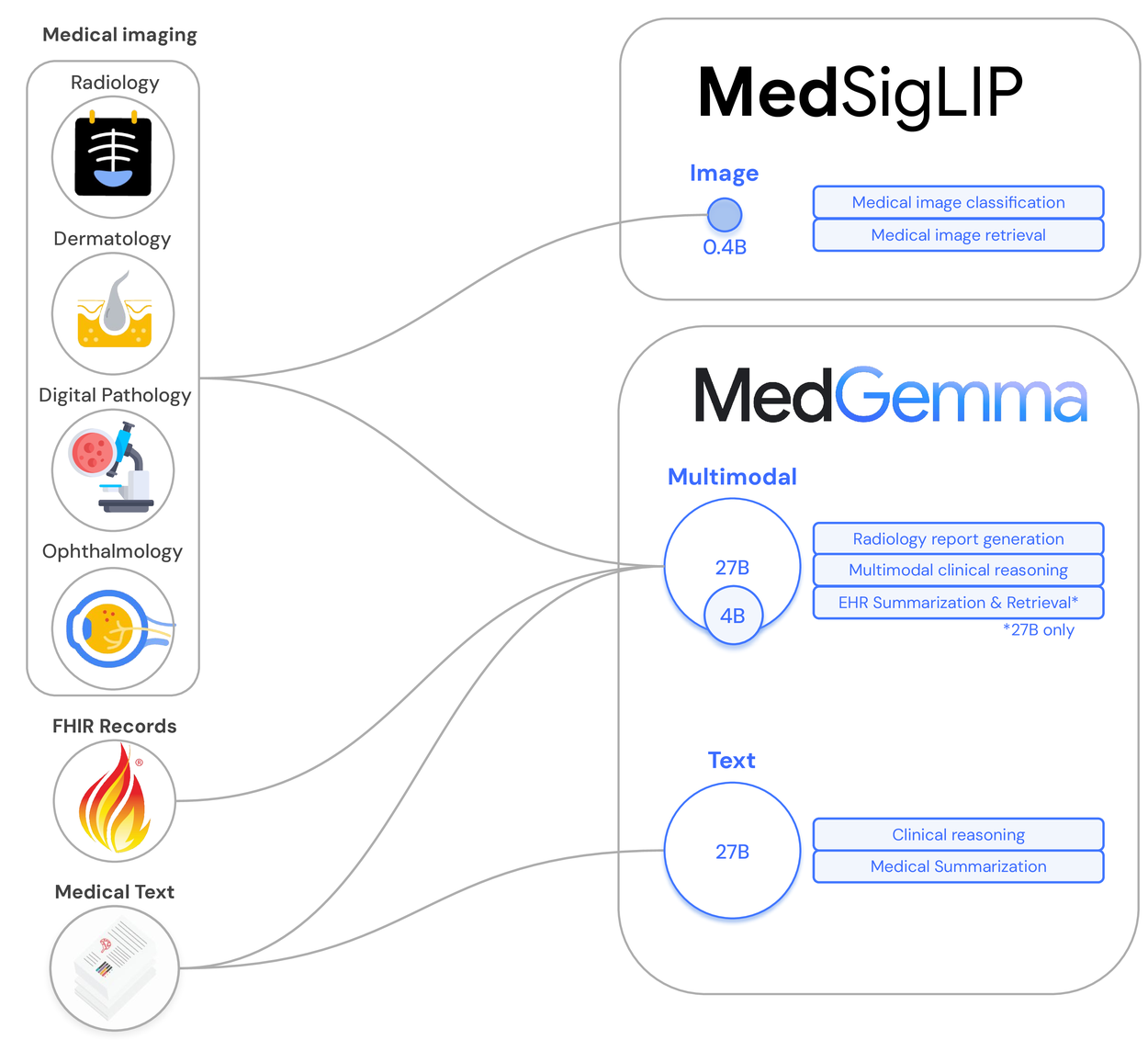
OpenMed 是一个由社区驱动的非营利性项目,专门为医疗保健和生命科学领域构建并分享可直接用于生产环境的人工智能模型。 该项目的核心使命是,让最强大的人工智能医疗工具不再被昂贵的商业壁垒所限制,而是免费提供给所有人使用。 OpenMed 致力于通过提供免费、强大且透明的工具,来加快相关领域的研究,为开发者提供支持,并最终改善患者的治疗结果。该项目所有的模型和工具都基于 Apache 2.0 许可协议发布,这意味着任何人都可以免费使用和修改。项目在Hugging Face平台上托管了数百个预训练模型,主要集中在“医疗与临床具名实体辨识”(NER)技术,能够识别和分类文本中的关键医疗信息,例如疾病、药物、解剖结构和基因组数据等。
功能列表
- 开源AI模型库:提供超过380个专门针对医疗和生命科学领域的预训练AI模型。
- 医疗具名实体辨识 (NER):提供一系列先进的NER模型,能够从非结构化文本(如临床笔记、研究论文)中自动识别和提取特定实体。
- 多样化的识别能力:NER模型覆盖多个专业领域,包括:
- 肿瘤学 (
OncologyDetect) - 病理学 (
PathologyDetect) - 药学 (
PharmaDetect) - 解剖学 (
AnatomyDetect) - 疾病 (
DiseaseDetect) - 基因组学 (
GenomeDetect)
- 肿瘤学 (
- 模型发现工具:提供一个名为
OpenMed NER Model Discovery的在线工具,帮助用户根据需求找到最适合的医疗和临床NER模型。 - 模型排行榜:建立并维护医疗AI模型的评估和基准测试排行榜,用于衡量和比较不同解决方案的性能。
- 社区驱动开发:作为一个社区项目,它汇集了众多开发者的贡献,并保持活跃的更新。
- 开放许可:所有模型和代码均在 Apache 2.0 许可下发布,确保了工具的免费和开放性。
使用帮助
OpenMed 的核心是其在 Hugging Face 平台上提供的众多预训练模型,这些模型无需复杂的安装流程,可以直接通过Hugging Face的 transformers Python库在任何支持Python的环境中调用和使用。
基本环境准备
要使用OpenMed模型,你的电脑需要安装Python和Hugging Face的 transformers 库。
- 安装 Python:如果你的系统中没有安装Python,请从官网
python.org下载并安装最新版本。 - 安装
transformers库:打开终端或命令行工具,输入以下命令来安装transformers库。如果你还需要使用PyTorch或TensorFlow作为后端,也请一并安装。pip install transformers torch # 或者如果你使用TensorFlow # pip install transformers tensorflow
如何操作和使用OpenMed模型
OpenMed 的主要功能是具名实体辨识(NER),即将一段文本中属于预设类别(如疾病、药物、身体部位)的词语识别出来。下面以一个具体的例子,说明如何使用 OpenMed/OpenMed-NER-DiseaseDetect-SuperMedical-355M 模型来识别文本中的疾病名称。
操作流程:
- 导入必要的工具首先,你需要从
transformers库中导入pipeline。这是一个高级辅助函数,可以让你轻松地使用Hugging Face上的任何模型。from transformers import pipeline - 加载指定的OpenMed模型使用
pipeline函数来加载模型。你需要为它指定两个核心参数:task: 对于OpenMed的实体识别模型,这个任务是"token-classification"或者"ner"。model: 模型的完整名称,你可以在OpenMed的Hugging Face主页上找到所有模型的名称。
# 初始化一个用于具名实体辨识的pipeline,并指定要使用的模型 ner_pipeline = pipeline( "token-classification", model="OpenMed/OpenMed-NER-DiseaseDetect-SuperMedical-355M" )当第一次运行这段代码时,
transformers库会自动从Hugging Face网站下载模型文件并缓存到你的本地电脑,过程可能需要一些时间,具体取决于你的网络速度和模型大小。 - 准备要分析的文本定义一个包含医疗信息的字符串。例如,一段描述患者症状的句子。
# 示例文本:一段包含疾病和症状描述的句子 text = "The patient was diagnosed with type 2 diabetes and hypertension. He was prescribed metformin." - 执行实体识别将准备好的文本输入到前面创建的
ner_pipeline中,它会返回一个包含所有识别出的实体的列表。# 运行模型进行实体识别 results = ner_pipeline(text) # 为了更好地查看结果,可以打印出来 print(results) - 解读识别结果程序的输出结果是一个列表,列表中的每个元素都是一个字典,代表一个被识别出的实体。每个字典包含以下关键信息:
entity_group: 实体的类别。对于这个疾病识别模型,类别就是Disease。score: 模型对于这个识别结果的置信度,分数越高代表模型越确定。word: 被识别出的具体词语或短语。start和end: 实体在原始文本中的起始和结束位置。
根据上面的示例文本,你可能会看到类似下面这样的输出:
[ {'entity_group': 'Disease', 'score': 0.998, 'word': 'type 2 diabetes', 'start': 29, 'end': 44}, {'entity_group': 'Disease', 'score': 0.999, 'word': 'hypertension', 'start': 49, 'end': 61} ]这个结果清晰地表明,模型成功识别出了文本中的两种疾病:“type 2 diabetes” 和 “hypertension”。
通过替换 model 参数,你可以用同样的方式调用OpenMed提供的其他任何NER模型,比如用于识别药物的 OpenMed/OpenMed-NER-PharmaDetect-SuperClinical-434M 或用于识别身体部位的 OpenMed/OpenMed-NER-AnatomyDetect-BigMed-278M。整个过程简单直接,让你能够快速地将先进的医疗AI功能集成到自己的应用程序或研究项目中。
应用场景
- 临床文档分析研究人员和医生可以利用OpenMed模型自动处理电子健康记录(EHR)、出院小结和病理报告。模型能快速从大量非结构化文本中提取关键信息,如诊断的疾病、使用的药物、患者的症状和相关的解剖学位置,从而辅助临床决策和加速病历回顾。
- 医学研究在科研文献分析中,研究者可以部署这些模型来扫描数千篇论文。模型能够自动识别和标记出与特定疾病、基因、药物或疗法相关的段落,帮助科研人员快速筛选信息,发现新的研究方向或进行文献综述。
- 药物警戒和安全监控制药公司和监管机构可以使用
PharmaDetect等模型来分析患者论坛、社交媒体或不良事件报告。通过自动识别提及特定药品和副作用的文本,可以更早地发现潜在的药物安全信号。 - 开发智能医疗应用开发者可以基于OpenMed的模型构建面向消费者的健康应用或服务。例如,开发一个能帮助患者理解化验报告的工具,该工具通过NER技术自动识别并解释报告中的医学术语。
QA
- OpenMed是什么?它收费吗?OpenMed是一个社区驱动的非营利项目,旨在提供开源、免费的医疗AI模型。 所有的模型和工具都基于Apache 2.0许可协议发布,因此完全免费,任何人都可以出于任何目的使用。
- 什么是具名实体辨识(NER)?具名实体辨识(NER)是人工智能领域的一项技术,它的作用是从文本中识别出具有特定意义的词语或短语(即“实体”),并将其划分为预先定义的类别。例如,在医疗文本中,NER可以将“高血压”识别为“疾病”,将“阿司匹林”识别为“药物”。
- 我需要很强的编程能力才能使用OpenMed的模型吗?不需要。借助Hugging Face的
pipeline工具,即使是编程初学者也能轻松加载和使用OpenMed的模型。如“使用帮助”部分所示,只需要几行简单的Python代码就可以完成模型的调用和实体识别。 - 这些模型的准确性如何?OpenMed项目非常注重模型的性能和准确性,并为此设立了公开的排行榜来进行评估和基准测试。 每个模型页面通常也会提供其在特定测试数据集上的性能指标,用户可以在使用前参考这些数据。